طراحی پرایمر با نرم افزار gene runner قسمت اول
نرم افزار Gene runner یک برنامه ساده ای است. کاربرد زیادی در مهندسی ژنتیک دارد. در سطح DNA و پروتئین میتوانیم آنالیزهای مربوطه را با این برنامه انجام دهیم و این امکان وجود دارد که توالی ها را هم بصورت دستی هم از سایت های اطلاعاتی مختلف در این نرم افزار وارد کنیم.
بطور خلاصه کارهایی که با این برنامه انجام میشود:
زمانیکه توالی DNA را وارد میکنیم برای یک توالی خاص و همچنین سایت های برش آنزیمی میتوانیم سرچ کنیم. در سطح پروتئین هم میتوان محل برش آنزیمها یا موتیف های خاص پروتئین را مشخص کرد. یکی از ساده ترین امکانات این نرم افزار تبدیل توالی DNA به پروتئین است. بنابراین خاصیت ترجمه هم دارد. از مزیت های نسبتا خوب این نرم افزار این است که نیازی به دسترسی به اینترنت ندارد و بصورت آفلاین قابل استفاده است.
مراحل کار با نرم افزار Gene runner
مرحله اول: نحوه وارد کردن توالی
در این نرم افزار بصورت default یک سری توالی ژن وجود دارد. که از گزینه فایل، روی open کلیک کنیم و از انجا توالی دلخواه را انتخاب و ok می کنیم. برای وارد کردن توالی مورد نظر خود از همان گزینه فایل، روی new کلیک کنیم و از بخش nucleic acid توالی خود را که کپی کرده بودیم ، اینجا paste میکنیم. برای وارد کردن توالی پروتئین نیز میتوان همین عمل را تکرار کرد فقط اینکه از گزینه فایل، وارد بخش پروتئین میشویم به جای nucleic acid.
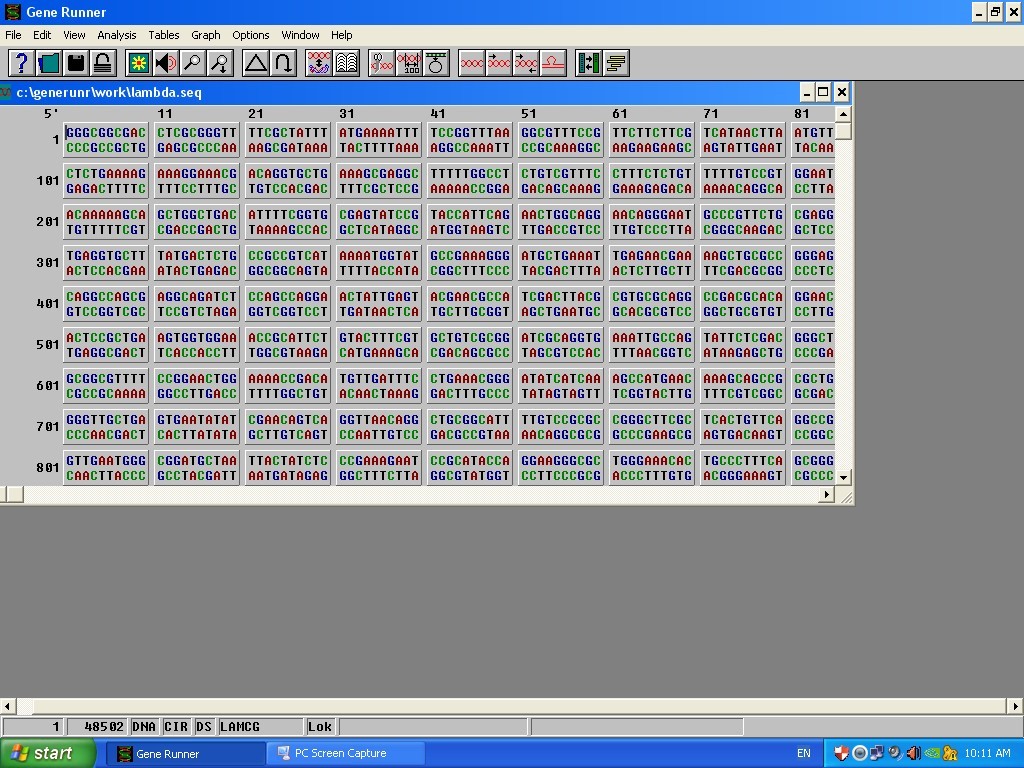
در اینجا ما توالی فاژ لامبدا را که در خود نرم افزار ذخیره شده بود را باز میکنیم و توالی را مشاهده میکنیم که هر نوکئیک اسید به رنگی نمایش داده شده است و این قابلیت را دارد که رنگ ها را تغییر داد. در همان نوار ابزار بالا روی شکل کلیک کرده و از منو باز شده میتوان رنگ ها را تغییر داد.
چون توالی فاژ لامبدا بسیار بزرگ است میتوان بخشی از توالی را کپی کرده (بطور مثال 2000 باز) و یک صفحه جدید از گزینه فایل بخش new را باز کرده و در آنجا paste کنیم. سپس این توالی را باید ذخیره کنیم تا در ادامه کار در دسترس داشته باشیم. برای ذخیره کردن از همان گزینه فایل، قسمت save as را زده و با اسم مشخصی آن را ذخیره میکنیم.
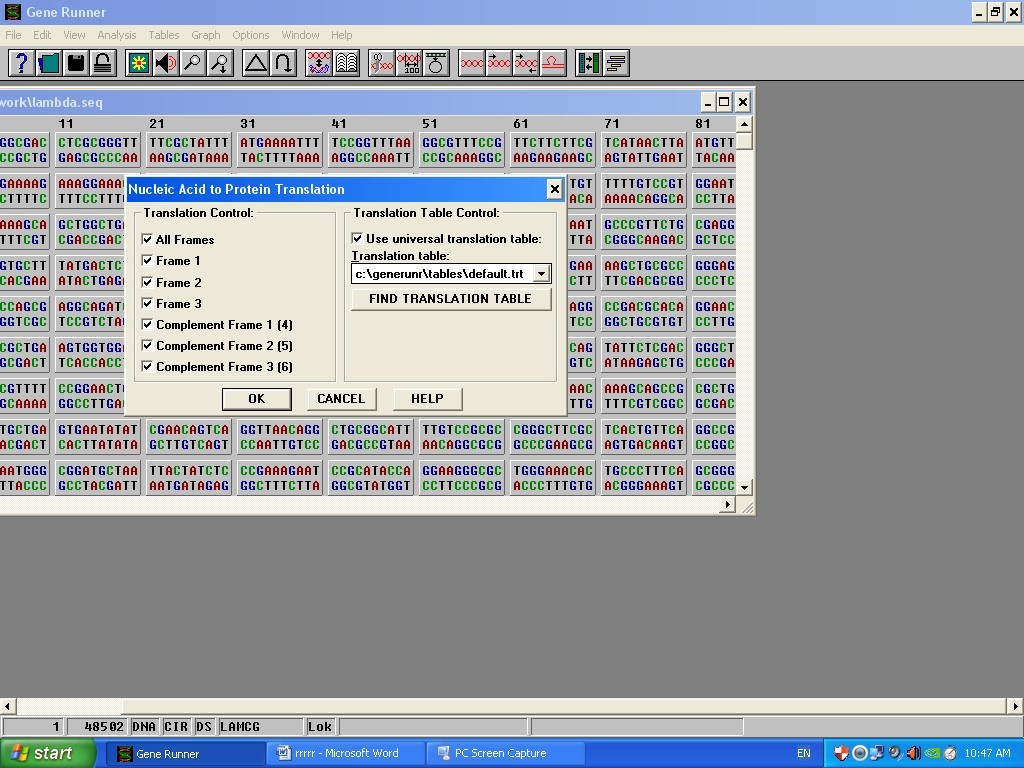
در گزینه view انچه که به درد ما میخورد translation میباشد. زمانیکه این گزینه را باز کنیم قادر است در تمام فریم ها توالی پروتئین را به ما بدهد و دارای 3 فرم سنس و 3 فریم انتی سنس میباشد.
*در همان نوار بالا یه علامتی به شکل وجود دارد، اگر بر روی آن کلیک نمائیم دیگر توالی پروتئین را به ما نشان نمیدهد.
سپس در منو باز شده مربوط به translation، یک فریم فقط تیک دار میکنیم و توالی پروتئینی را در یک فایل جدید به همان روش گفته شده در بالا ذخیره میکنیم.
از دیگر کارایی های خوب این نرم افزار آنالیز سایت های آنزیمی میباشد. یعنی اینکه ما قطعه ای داریم و میخواهیم بدانیم با چه انزیمهای بریده میشود و حتی بر حسب نیاز ما آنزیمهای خاصی را نیز میتواند معرفی کند.
بطور کلی میتوان توالی DNA را برای وجود سایت های برش انزیمی مشخص کرد. از 2 راه میتوان این عمل را انجام داد :
- با استفاده شکل قیچی که در نوار ابزار بالای نرم افزار وجود دارد.
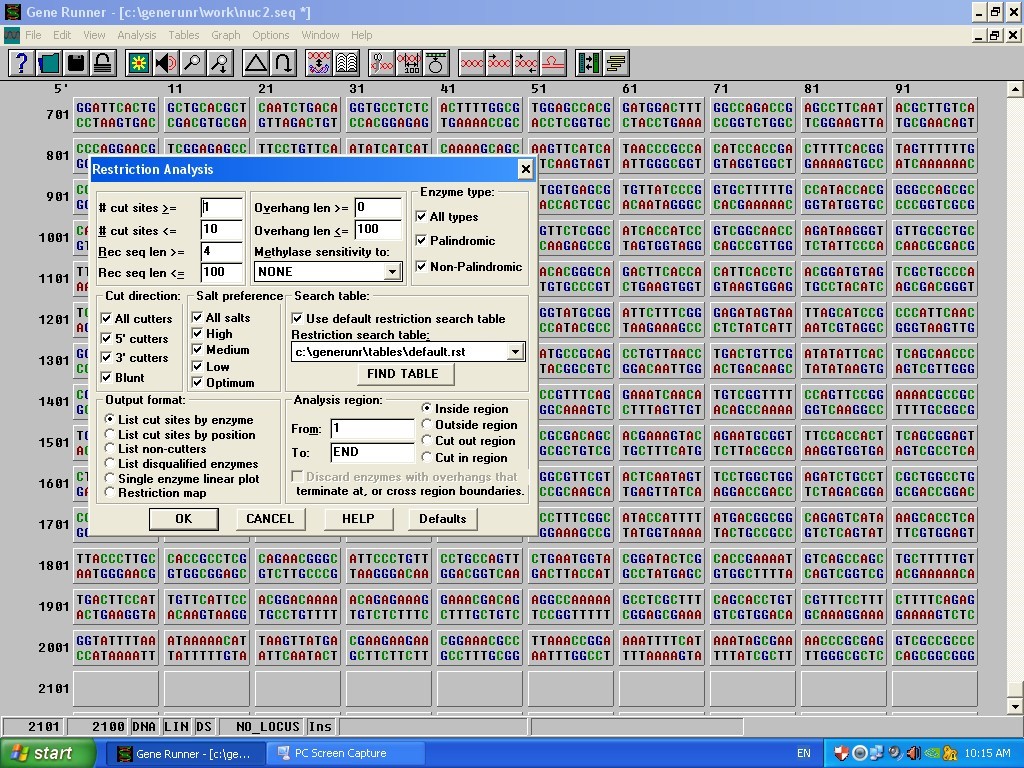
- در گزینه آنالیز، nucleic acid، بعد از آن restriction site را انتخاب میکنیم و بصورت جدول تمام انزیم ها و تعداد برش را به ترتیب نمایش میدهد.
از گزینه آنالیز، گزینه nucleic acid، بعد از آن restriction site انتخاب کرده، یک منو باز میشود که در بخش output format، گزینه list cut by enzymes بصورت default تیک دار است و سپس ok را می زنیم و لیستی از تمام انزیمها را نشان میدهد.
جایگاه انزیم ها را روی توالی نیز میتوان پیدا کرد، اگر روی یک یا چند آنزیم در جدول دابل کلیک کنیم بطوری که علامت ستاره جلوی نامشان قرار گیرد، سپس در همان صفحه ای که باز شده اگر روی گزینه highlight کلیک کنیم روی توالی جایگاه برش را نشان میدهد.
در همین صفحه اگر روی گزینه map کلیک کنیم نقشه شماتیکی دیگری هم از محل برش را می بینیم.
در صفحه مربوط به انزیم های محدود کننده، بخش output format اگر به جای list cut by enzymes، گزینه single enzyme linear plot را تیک دار و سپس ok کنیم، محل برش را بصورت خطی نشان میدهد.
تعاریف:
- Cut position
- Inside region
- Cut in region
- Outside region
- Cut out region
این موارد در همان بخش آنالیز، گزینه nucleic acid، بعد از آن restriction site است که در صفحه باز شده، قسمتی به نام cut position وجود دارد و 4 ایتم بالا را شامل میشود.
Cut in region: زمانیکه این گزینه را تیک دار میشود، یعنی اینکه به دنبال انزیم های میگردد. که فقط در ناحیه ای انتخاب شده وجود دارند و به قبل و بعد از آن ناحیه کاری ندارد.
به این صورت است که در همان توالیDNA کپی شده، یک بخشی از ان را هایلات میکنیم و سپس در گزینه انالیز ،زمانی که سایت های برش را انتخاب میکنیم از منو باز شده گزینه single enzyme linear plotو cut in region را تیک دار کرده و ok میکنیم. در نتیجه نقشه برش آنزیم ها را بصورت خطی تنها در بخش هایلایت شده نشان میدهد.
Inside region: زمانی که این گزینه تیک دار میشود؛ به دنبال آنزیم های که در این ناحیه هایلات شده. و همچنین اطراف آن وجود دارند، میگردد و ان را نمایش میدهد.
تفاوت این دو مورد در این است که در cut in تنها آنزیمهای که در این ناحیه است را نشان میدهد. اما inside آنزیم های داخل و همچنین در خارج از توالی مورد نظر را نیز نشان میدهد. که برای تایید حضور ژن در پلازمید و خود پلازمید کاربرد دارند.
Cut out region: این گزینه نیز به محل برش انزیمهای که در خارج از ناحیه هایلات شده است؛ مربوط میشود.
Outside region: به دنبال انزیمهای که در خارج از توالی مورد نظر و داخل ان وجود دارند، میباشد.
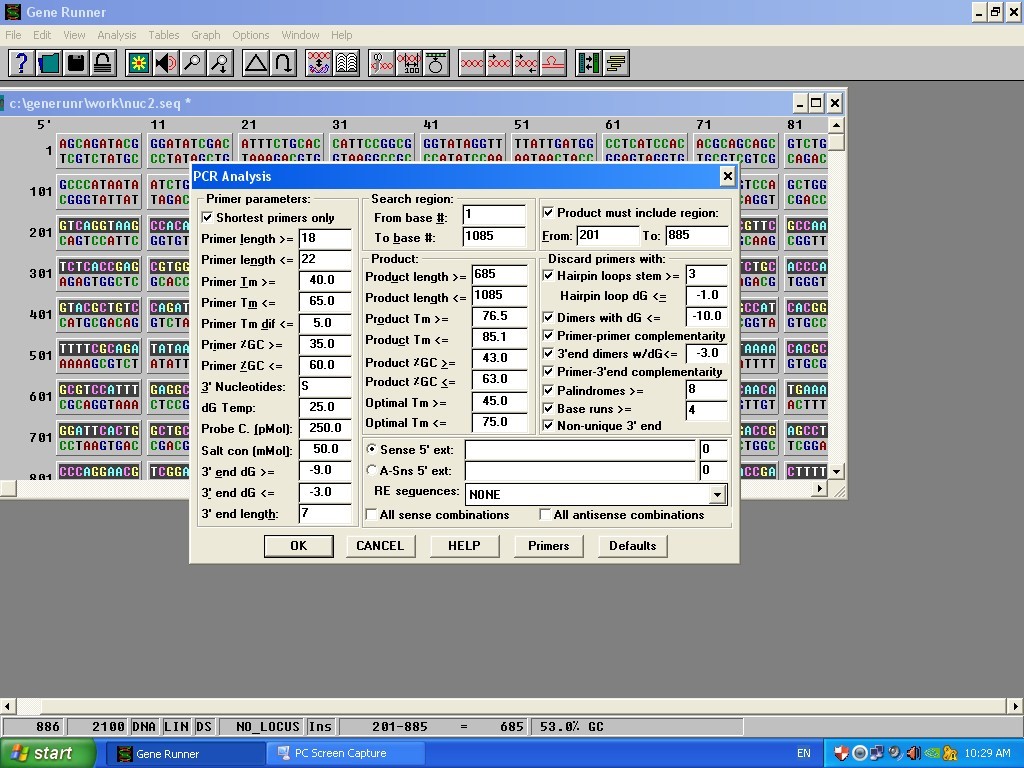
کاربرد دیگر نرم افزار برای طراحی پرایمر است که براساس target base میباشد. برخلاف سایر نرم افزارهای دیگر که براساس primer base است. یعنی براساس توالی که به آن داده شده است پرایمر را نمایش میدهد.
این نرم افزار Tm محصول و پرایمر را محاسبه میکند و براساس آن یک پرایمر بهینه با یک Tm بهینه به ما میدهد و Tm محصول را نیز در محاسبه می گنجاند.
فرمولی را که برای محاسبه Tm بهینه استفاده میکند بصورت زیر است:
(0.3Tm primer + 0.7Tm product ) *14.9
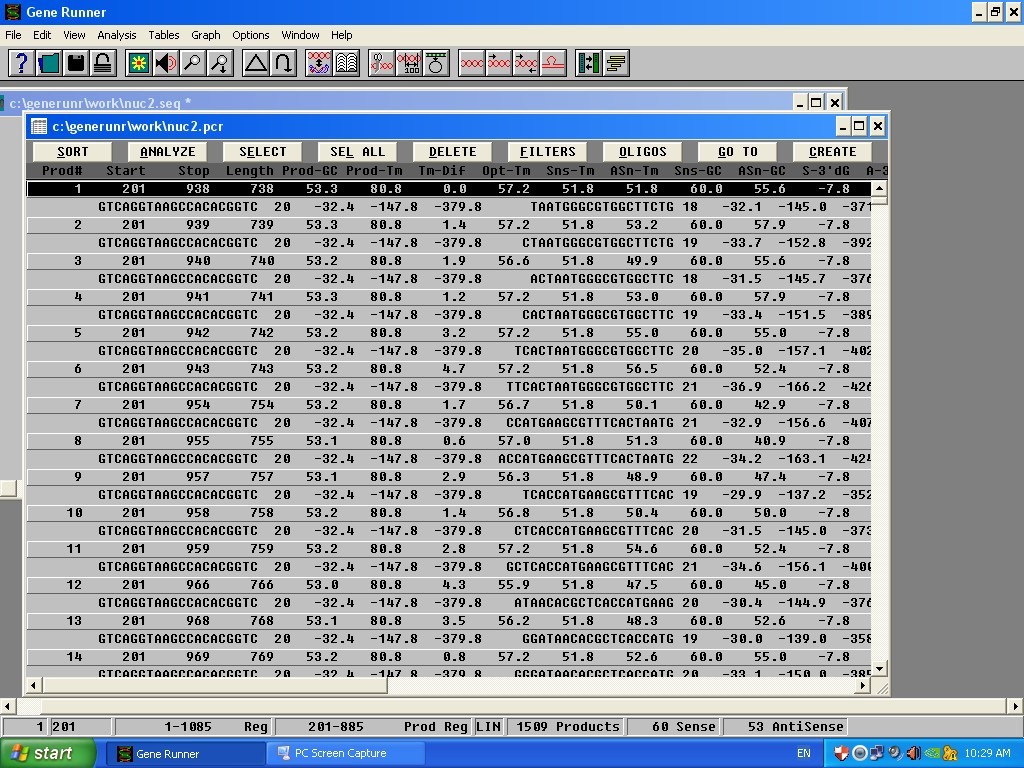
در گزینه آنالیز، primer PCR را انتخاب کرده و لیست تمام پرایمرها را به ترتیب از بهترین پرایمر نمایش میدهد.
روی اولین پرایمر کلیک کرده ( که هم سنس هم انتی سنس وجود دارد) سپس از همان نوار ابزار بالا در گزینه آنالیز ، oligo را میزنیم و تمام اطلاعات مربوط به پرایمر را نشان میدهد و در همان منو باز شده اگر switch oligo را تیک دار کنیم رشته مقابل را به ما نشان میدهد.
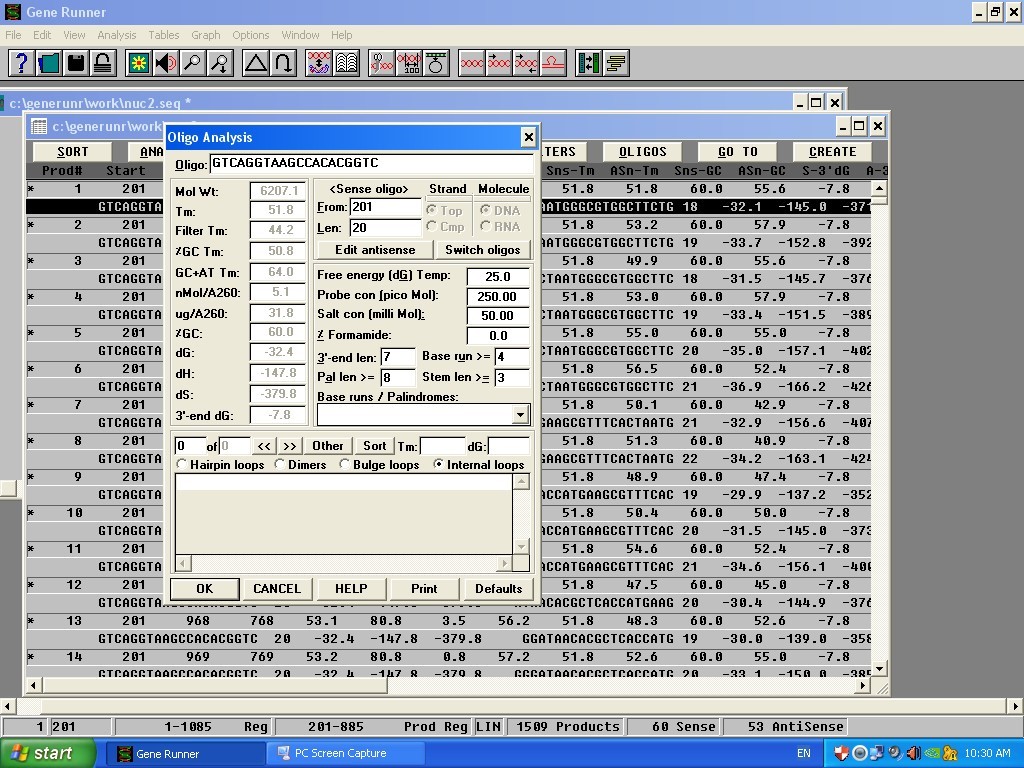 در این منو گزینه هایی نیز که بیان کننده وجود ساختارهای ثانویه از جمله hairpin loop, dimer, bulge loop, internal loop وجود دارد. که جز برتری های این نرم افزار محسوب میشود.
در این منو گزینه هایی نیز که بیان کننده وجود ساختارهای ثانویه از جمله hairpin loop, dimer, bulge loop, internal loop وجود دارد. که جز برتری های این نرم افزار محسوب میشود.
اطلاعات مربوط به این ساختارها را از جمله Tm و dG را نیز باید به دقت بررسی شود.
قبل از هر چیزی باید از یک سری ویژگیهای که برای پرایمر ضروری است ، اطلاع داشت. از جمله : وجود GC clamp در انتهای پرایمر برای اینکه اتصال به الگو محکم باشد.
درصد CG هم مهم است بهتر است 50 به 50 باشد اما یک قانون نیست.
dG یا همان انرژی ازاد هم مهم است. زمانیکه صفر است یعنی واکنش در حال تعادل است و تعداد پرایمرهای متصل با آزاد برابر است. زمانیکه منفی میباشد یعنی تعداد پرایمرهای متصل بیشتر است و مثبت بودن یعنی واکنش به خوبی انجام نمیشود.
انتهای 3‘ نیز عامل مهمی است.مانند dG هر چه منفی تر باشد بهتر است و اگر خیلی منفی هم باشد باعث ایجاد mispriming میشود.
علاوه بر پرایمرهای را که خود نرم افزار به ما میدهد خودمان نیز میتوانیم از گزینه آنالیز، oligo را انتخاب کرده و در منو باز شده به جای توالی در کادر بالا توالی مورد نظر خود را دستی وارد کنیم و سپس تمام ویژگیهای پرایمر را بررسی کنیم. در این حالت ساختارهای ثانویه حتما باید بررسی شوند در صورت وجود ساختاری که dG نزدیکی به dG پرایمر داشته باشد یعنی اینکه احتمال ایجاد این ساختار در پرایمر بالا میباشد و باید پرایمر را طوری دیگر طراحی کرد.





اشتراک گذاری