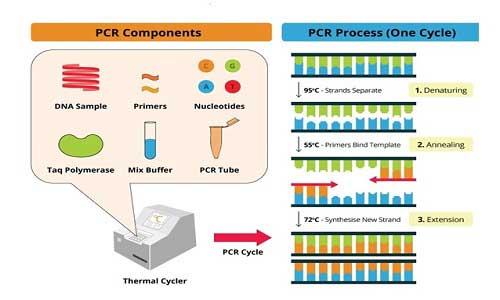
پروتوکل کلاسیک PCR
پروتوکل کلاسیک PCR یک نقطه شروع عمومی برای انجام PCR با استفاده از DNA ژنومی یا cDNA به عنوان یک الگو ارائه می دهد. همانطور که گفته شد پروتکل برای یک حجم واکنش 50 میکرولیتر طراحی شده است. با این حال ، واکنش را می توان تا 100 میکرولیتر یا پایین تر و تا 20 میکرو لیتر کوچکتر کرد.
واکنش PCR نسبت به آلودگی DNA دیگربسیار حساس است. بنابراین ، باید احتیاط کرد تا مطمئن شوید فضایی که واکنش PCR در آن انجام می شود یا اجزای مورد استفاده در واکنش PCR تمیز و فضا عاری از گرد و غبار است (کابینت PCR بسیار توصیه می شود) ، و تا آنجا که ممکن است برای کاهش احتمال آلودگی ، از پوشیدن دستکش استفاده کنید ، فقط از پیپترهای اختصاصی برای ساخت مخلوط و اجزای واکنش PCR استفاده کنید.
متغیرهای متعددی وجود دارد که می توانند بر نتیجه واکنش PCR تأثیر بگذارند. پروتکل زیر استفاده از پلیمراز hot-start Taq DNA polymeras استفاده می کند (آنزیمی که لازم نیست آنزیم در ابتدای واکنش اضافه کنید بلکه می تواند آن را به مستر میکس اضافه کنید) ، یک بافر PCR که حاوی MgCl2 نیست ، و غلظت معرف ها که نشان دهنده یک نقطه شروع خوب برای بهینه سازی اختصاصی آغازگر است. پارامترهایی که می توانند در یک واکنش خاص به همراه دامنه های قابل قبول تنظیم شوند ، در جدول واکنش استاندارد ذکر شده است.
چرخه حرارتی PCR دارای سه پارامتر است که می توان آنها را دستکاری کرد: زمان ، دما و میزان سرعت چرخش دستگاه بین دما (سرعت رمپ). شرایط مطلوب برای یک واکنش معین به نوع دستگاه که در آن واکنش اجرا خواهد شد ، درجه حرارت ذوب آغازگرهای مورد استفاده ، اندازه قطعه مورد نظر برای تکثیر و ماهیت توالی الگو(محتوایGC، ساختار ثانویه و…) و ظرفی که مخلوط واکنش در آن تکثیر می شود، بستگی دارد.
برای بهینه سازی بیشتر شرایط واکنش PCR می توان از نرخ رمپ استفاده کرد. برای اکثر واکنشهای PCR ، نرخ رمپ به تنظیم پیش فرض کارخانه سازنده (اغلب تنظیمات حداکثر) تنظیم می شود تا سریعاً بین دمای دناتوراسیون ، annealing و elongation و سرعت بخشیدن به سرعت کلی PCR انجام شود. معمولاً تنظیم این پارامتر ضروری نیست. با این حال ، برای جبران کاهش زمان شروع ، برای مثال ، تنظیم نرخ رمپ ممکن است دامنه چرخه را برای تکثیرموفقیت آمیز توالی هدف افزایش دهد. علاوه بر این ، هنگامی که درجه حرارت annealing نسبتاً بالا است (به عنوان مثال ، >60) ، elongation می تواند به اندازه کافی کارآمد باشد که درجه حرارت را برای مرحله elongation تغییر ندهد ، و این منجر به PCR دو مرحله ای می شود که می تواند زمان کلی چرخه را کاهش دهد. یک مرحله رمپینگ از بین می رود. این صرفه جویی در زمان حداقل است از آنجایی که دامنه کلی ازannealing تا elongation تا دناتوراسیون به طور موثر همان اختلاف دما را به عنوان دو مرحله از annealing / elongation تا دناتوراسیون پوشش می دهد.
| Master mix for 10 tubes (prepare for 12 tubes) | Per tube volume | Final concentration | Components |
| 60 μl | 5 μl | 1× | 10× PCR buffer MgCl2-free |
| 36 μl | 3 μl | 1.5 mM | 25 mM MgCl2a |
| 12 μl | 1 μl | 0.2 mM each dNTP | 40 mM dNTP mixb |
| 60 μl | 5 μl | 1 μMc | 10 μM forward primer |
| 60 μl | 5 μl | 1 μMc | 10 μM reverse primer |
| 369 μl | 30.75 μl (to a final volume of 50 μl) | Sterile, nuclease-free H2O | |
| 3μl | 0.25 μl | 0.025 U/μl | 5 U/μl hot-start Taq DNApolymerased |
| — | 1μl | 2 ng/μl | >100 ng/μl templatee |
| a. غلظت MgCl2 را می توان برای بهینه سازی واکنش PCR تیترکرد. غلظت MgCl2 در اکثر موارد بین 1.5 تا 4.0 میلی مولار است. b . تعداد زیادی از مخلوط های تجاری dNTP در دسترس هستند. بیشتر آنها 40 میلی م.لار می باشند (10 میلی مولار از هر 4 dNTP) است. c. غلظت Primer همچنین می تواند برای بهینه سازی واکنش PCR تیتر شود. غلظت آغازگر نهایی به طور کلی می تواند دردامنه 0.2 تا 1 میکرومولارتنظیم شود. d. غلظت آنزیم متفاوت خواهد بود. در صورتی که از پلیمراز غیر hot-start استفاده می کنید، آنزیم نباید به میکروتیوب واکنش اضافه شود تا زمانی که لوله ها در چرخه حرارتی 95 درجه سانتیگراد قرار بگیرند تا از شکل گیری محصولات غیر اختصاصی خودداری شود. e. غلظت DNA الگو می تواند متفاوت باشد ، اما به طور کلی غلظت نهایی DNA الگو از 10 نانوگرم در میکرولیتر تجاوز نمی کند. این روش به اندازه کافی حساس است تا بتواند مقادیر پیکومول از الگو را تشخیص دهد. |
آماده سازی مخلوط واکنش
1. سطح کار را با محلول سفید کننده 10٪ پاک کنید.
2- اجزای PCR را روی یخ ذوب کنید. پس از اینکه کاملاً ذوب شد، قبل از استفاده ، هر یک را بطور ملایم ورتکس کنید. توجه داشته باشید که برخی از پلیمرازها می توانند به ورتکس بسیار شدید حساس باشند. بنابراین بهتر است که DNA پلیمراز را با زدن انگشت به لوله مخلوط کنید تا محتویات را مخلوط کرده و سپس آن را بی درنگ روی یخ برگردانید.
3. مطابق جدول واکنش استاندارد (جدول 10.2.5) یک ترکیب مستر روی یخ تهیه کنید. به جز الگو ، هر معرف را به ترتیب ذکر شده در جدول اضافه کنید. مخلوط واکنش را سریع و آرام ورتکس کنید. معمولا حجم مستر میکس ابتدایی برای یک آزمایش PCR داده شده ، برای هر جفت آغازگر ،به تعداد n لوله + 2 است (به عنوان مثال ، به جای 10 لوله، یک ترکیب اصلی تهیه کنید برای 12 نمونه) این روش مشکل خطا در پیپتینگ را رفع می کند. علاوه بر این ،مستر میکس باقی مانده میتواند بدون الگو در یک لوله جداگانه به عنوان یک کنترل منفی برای واکنش استفاده شود. هرگونه تکثیری که در این لوله مشاهده شود می تواند نشانه ای از آلوده بودن ترکیب واکنش ونیاز به آزمایش مجدد باشد.
الگوی DNA به دلایل زیر در این مرحله کنار گذاشته شده است:
(1) این فرصت یکسان برای همه نمونه ها، تا سازگاری با میکس یکسانی ایجاد شود.
(2) یک کنترل منفی (نمونه ای که شامل الگو نیست و بنابراین نباید محصول را تکثیرکند) برای ترکیب کلی واکنش می تواند برای بررسی آلودگی استفاده کرد.
4- مقدار 49 میکرولیتر از مستر میکس را درون هر لوله PCR با دیواره نازک مخلوط کنید و سپس 1 میکرولیتر از الگو را به هر لوله اضافه کنید. لوله ها را روی یخ نگه دارید. لوله ها را برای وجود حباب یا مواد باقی مانده در دیواره لوله ، بررسی کنید. ایده خوبی است که به سرعت ورتکس و سانتریفیوژ انجام شود تا اطمینان حاصل شود که تمام معرف ها مخلوط شده و در پایین قرار دارند.
5- در صورتی که دستگاه درب گرم کن ندارد، 25 تا 50 میکرولیترروغن معدنی استریل و بدون نوکلئازرا به هر لوله اضافه کنید. اگر دستگاه دارای یک درب گرم است ، حتماً درب را طوری تنظیم کنید که به طور محکم در بالای لوله های واکنش قرار گیرد. به عنوان گزینه ای جایگزین برای روغن معدنی ، از پارافین استفاده می شود.


اشتراک گذاری